β-アミラーゼの構造・機能解析

β-アミラーゼは、デンプンの非還元性末端からマルトース(麦芽糖)単位でα-1,4グリコシド結合を逐次分解するエキソ型の酵素であり、生成物はβ-アノマーのマルトースを生成する。β-アミラーゼはアミロースをほぼ分解することができるが、アミロペクチンを基質とした場合、α-1,6結合の手前で反応が止まりβ-リミットデキストリンが残る。加工デンプン工場では、α-アミラーゼでデンプンを液化糖にし、β-アミラーゼやプルラナーゼを作用させることによって、高濃度のマルトースシェロップ(水飴)を得ている。
多くのアミラーゼがα-アミラーゼと一次構造や機能の上で類似性を持つのにたいし、β-アミラーゼはα-アミラーゼとそのような類似性がなく、アミラーゼの中で独自の位置を占めている。 近年、いくつかの植物および微生物由来のβ-アミラーゼの構造が明らかになったが、その触媒メカニズムは、グルコアミラーゼと同様に反転型(inverting)酵素であるが、まだ多くのことがわかっていない。そこでX線結晶構造解析および反応速度論的解析を駆使することでβ-アミラーゼの詳細な触媒メカニズムの解明を目的とする。
β-アミラーゼとは?

沈殿剤にPEGと硫安を用いることで図1のような結晶を得ることができる。この結晶を振動させながらX線を露光することで、それぞれの振動角ごとの反射データを得る。これらの回折データをコンピュータを用いて計算することで、コンピュータグラフィックス上に分子モデルを表示させることができる。
β-アミラーゼの結晶


図1 β-アミラーゼの結晶
図2は全体の構造を示している。β-アミラーゼは3つのドメインから構成され、緑色の部分は、触媒ドメインを示し、(β/α)8バレル構造を形成している。また青色の部分は、デンプン結合ドメインで逆平行なβシートで構成されている。黄色と赤色のスティクモデルは結合してるマルトオリゴ糖を示している。
全体構造

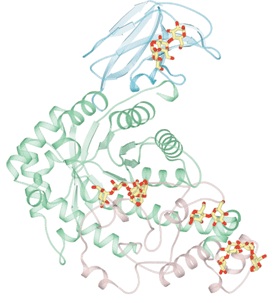
図2 β-アミラーゼの全体構造
緑色は、ドメインAで触媒ドメイン。マゼンタはドメインB。青色はドメインCでデンプン結合ドメイン。黄色と赤色のスティックモデルはマルトオリゴ糖。
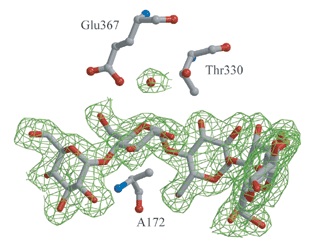
この酵素の触媒残基は、Glu172とGlu367であることがこれまでの研究で明らかになっている。左の図は、Glu172をAlaに置換した変異酵素と基質との複合体の電子密度図である(図3)。基質は左から2つ目と3つ目のグルコース残基の間で大きくねじれて活性部位に結合することがわかった。またグルコシド結合の切断直後に攻撃すると予想される水分子も観測されている。
活性部位に結合している糖の電子密度図

図3 活性部位に結合している糖の電子密度図
緑色は、電子密度。スティックモデルは触媒に関与するアミノ酸残基と活性部位に結合している糖。
どちらの触媒残基が塩基触媒として働くかを調べるためにどちらか一方の触媒残基をAlaのような側差の短いアミノ酸残基に置換し、求核試薬を加えることで活性が回復するかどうか調べた結果、Glu367をAlaに置換したものだけが活性の回復を示した。また先程のX線結晶解析の結果とwild酵素とプロダクトであるマルトースとの複合体の結果を重ねて考察すると、Glu172の側差の酸素原子の不対電子の軌道の向きが切断されるグルコシド結合の酸素原子の方へ向いている。従って本酵素の触媒機構は、まず、プロトンドナーであるGlu172によって切断されるグルコシド結合の酸素原子にプロトンを供与し、塩基触媒残基であるGlu367によって C1炭素付近にある水分子を求核攻撃し、活性化された水分子がC1炭素に付加して、β-マルトースを生成すると考えられる(図4)。
触媒メカニズム

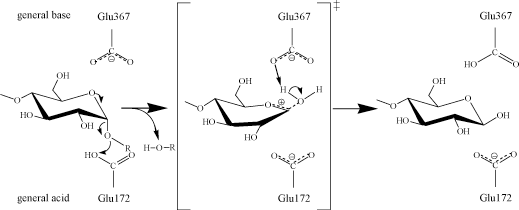
図4 β-アミラーゼの触媒メカニズム
図5は、wild酵素とマルトースとの複合体(灰色)とこれまで報告されているβ-アミラーゼの複合体の活性部位を比較したステレオ図である。Glc1は非還元末端でGlc1とGlc2はどの複合体においてもほとんど同じオリエンテーションをとる。しかしながら、b)と c)においてGlc3からのオリエンテーションの違いが見られた。マルトースとの複合体において、Glc3にグルコース残基が占有するとThr330 からのアミノ酸残基が座標シフトすることで側差の酸素原子とGlc3の2位の水酸基との間で水素結合を形成すると考えられる。しかしながら、b)のE172Aとマルトペンタオースとの複合体(黄色)はGlc3にグルコース残基が占有しているのにもかかわらずThr330からのアミノ酸残基の座標シフトは観測されなかった。おそらく基質が加水分解される直前まで、図3で見られるように触媒残基のGlc367とThr330の側差の酸素原子で水分子を安定に保持するのが重要であると考えられる。
他の複合体の活性部位の比較

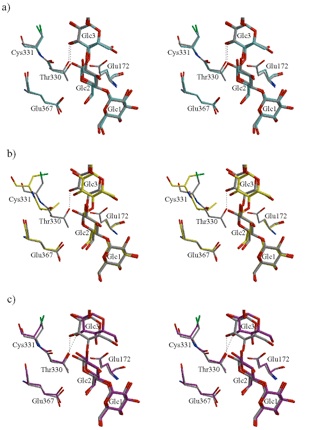
図5 活性部位の比較
マルトースとの複合体(灰色)とa)大豆β-アミラーゼとマルトースとの複合体(水色) b)E172Aとマルトペンタオースとの複合体(黄色) c)他の微生物由来β-アミラーゼとマルトースとの複合体(紫色)
図6は、ドメインCと糖が結合している状態を示している。雲のようにもこもことしているものは分子表面である。糖の2つのグルコース残基と2つのTrpが分子表面でスタックしている様子が分かる。このような結合はCGTaseのドメインEやグルコアミラーゼのドメインCにも見られる。
ドメインCの糖質結合部位

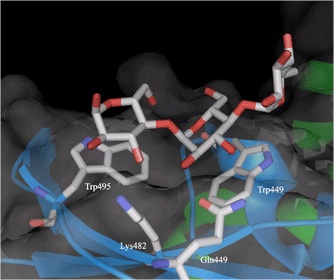
図6 ドメインCの糖質結合部位
図7は、ドメインBに糖が結合している様子を示した図である。左下に見える糖は活性部位に結合している糖の還元末端側です。以前まではSite 2にしか確認できなかったが、触媒残基に変異を導入することでSite 3にも糖が結合してるのを観測した。
ドメインBの糖質結合部位

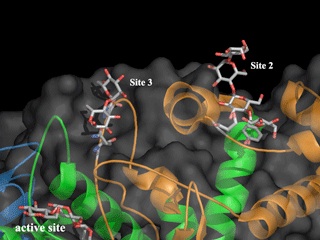
図7 ドメインBの糖質結合部位
図8は、a)は活性部位が中心に見える方向の分子表面図で、b)はa)をy軸に180度回転させたときの分子表面図である。すべての糖結合部位は、活性部位の方向にある。おそらく本酵素は、活性部位のある分子表面で基質であるデンプンを捕らえ効率よく加水分解反応を行うと考えられる。
糖結合部位について

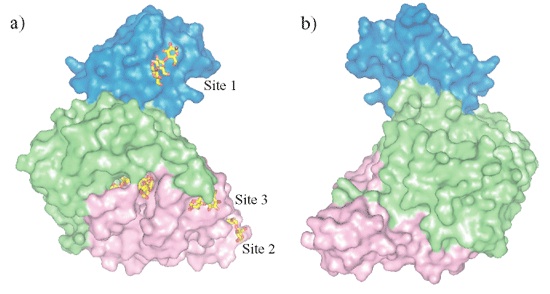
図8 糖質結合部位
図9の横軸をphi、縦軸をpsiとし、図中の実線の等高線は、マルトースのMM3の iso-energyを示している。等高線の中心が最も安定な領域である。●は、E172Aとマルトペンタオースとの複合体を示し、△□は、大豆β-アミラーゼで生成物であるマルトースを用いた時に、縮合反応によって生じたマルトテトラオースとの複合体を示している。Glc2とGlc3の間のグルコシド結合を除き、他のグルコシド結合は安定なcis型に近い値を取っている。基質は、Glc2とGlc3の間ねじれており、この間のマルトース単位のコンフォメーション エネルギーは、安定なcis型の時よりも、20 kcal/mol高い。大豆βアミラーゼの場合も同じような値をとっている。従って、Glc2とGlc3の間のグルコシド結合は、基質が結合することによって無理な構造を強いられており、 β-アミラーゼは、ES複合体の状態で、切断されるグルコシド結合を不安定化させ、高いエネルギーレベルとすることで、この段階と遷移状態の間のエネルギーの差 を減少させ、迅速な水解速度を生じていると推定される。
なぜ基質は容易に加水分解されるか?

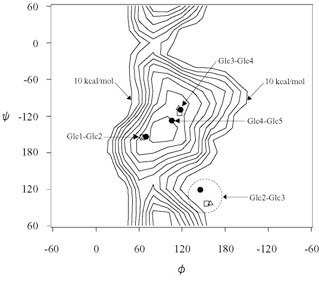
図9 マルトースのMM3の iso-energy
copyright (c) 2004-2011 Miyake's HP All rights reserved.
<文献>
新田康則, 三宅英雄 "微生物β-アミラーゼの構造と機能" 『食品酵素化学の最新技術と応用—フードプロテオミクスへの展望』2章1節, シーエムシー出版, p12-20. (2004)
Ye, Z, Miyake, H., Tatsumi, M., Nishimura. S., Nitta, Y. “Two additional carbohydrate-binding sites of beta-amylase from Bacillus cereus var. mycoides are involved in hydrolysis and raw starch-binding.” J. Biochem., 135(3), 355-363. (2004)
Miyake, H., Kurisu, G., Kusunoki, M., Nishimura, S., Kitamura, S., Nitta, Y. “Crystal Structure of a Catalytic Site Mutant of β-Amylase from Bacillus cereus var. mycoides Cocrystallized with Maltopentaose.” Biochemistry, 42(19), 5574-581. (2003)
Oyama, T., Miyake, H., Kusunoki, M., Nitta, Y. “Crystal Structures of β-Amylase from Bacillus cereus var. mycoides in Complexes with Substrate Analogs and Affinity-Labeling Reagents.” J. Biochem., 133(4), 467-474. (2003)
Miyake, H., Otsuka, C., Nishimura, S., Nitta, Y. “Catalytic mechanism of β-amylase from Bacillus cereus var. mycoides: chemical rescue of hydrolytic activity for a catalytic site mutant (Glu367-->Ala) by azide.” J. Biochem., 131(4), 587-591. (2002)